- W&B Run を初期化し、再現性のために Run に関連付けられたすべての設定(configs)を同期する。
- MONAI transform API:
- 辞書形式データのための MONAI Transforms。
- MONAI
transformsAPI に従って新しい transform を定義する方法。 - データ拡張のために強度(intensity)をランダムに調整する方法。
- データの読み込みと可視化:
- メタデータを含む
Nifti画像の読み込み、画像のリストの読み込みとスタック。 - トレーニングと検証を加速するための IO と transform のキャッシュ。
wandb.Tableと W&B 上のインタラクティブなセグメンテーションオーバーレイを使用したデータの可視化。
- メタデータを含む
- 3D
SegResNetモデル のトレーニング- MONAI の
networks、losses、metricsAPI の使用。 - PyTorch トレーニングループを使用した 3D
SegResNetモデル のトレーニング。 - W&B を使用したトレーニング 実験 の追跡。
- モデルのチェックポイントを W&B 上の モデルアーティファクト として ログ および バージョン管理 する。
- MONAI の
wandb.Tableと W&B 上のインタラクティブなセグメンテーションオーバーレイを使用して、検証 データセット に対する 予測 を可視化し比較する。
セットアップとインストール
まず、最新バージョンの MONAI と W&B をインストールします。W&B Run の初期化
新しい W&B Run を開始して 実験 の追跡を開始します。適切な設定システム(config system)を使用することは、再現可能な 機械学習 のための推奨されるベストプラクティスです。 W&B を使用して、すべての 実験 の ハイパーパラメーター を追跡できます。データの読み込みと変換
ここでは、monai.transforms API を使用して、マルチクラスのラベルを one-hot 形式のマルチラベルセグメンテーションタスクに変換するカスタム transform を作成します。
データセット
この 実験 で使用される データセット は http://medicaldecathlon.com/ から提供されています。これは、マルチモーダル・マルチサイトの MRI データ(FLAIR, T1w, T1gd, T2w)を使用して、神経膠腫(Gliomas)、壊死/活動性腫瘍、および浮腫をセグメント化します。 データセット は 750 個の 4D ボリューム(トレーニング用 484 + テスト用 266)で構成されています。DecathlonDataset を使用して、 データセット を自動的にダウンロードおよび解凍します。これは MONAI の CacheDataset を継承しており、メモリサイズに応じて cache_num=N を設定してトレーニング用に N 個のアイテムをキャッシュしたり、デフォルトの 引数 を使用して検証用のすべてのアイテムをキャッシュしたりできます。
注:
train_dataset に train_transform を適用する代わりに、トレーニングと検証の両方の データセット に val_transform を適用します。これは、 トレーニング を開始する前に、両方の分割(split)からサンプルを可視化するためです。データセット の可視化
W&B は画像、ビデオ、オーディオなどをサポートしています。リッチメディアを ログ に記録して、結果を探索したり、 Run 、 モデル 、 データセット を視覚的に比較したりできます。セグメンテーションマスクオーバーレイシステム を使用して、データのボリュームを可視化します。セグメンテーションマスクを テーブル (Tables) に ログ 記録するには、テーブルの各行に対してwandb.Image オブジェクトを提供する必要があります。
疑似コードの例を以下に示します。
wandb.Table オブジェクト、および関連する メタデータ を受け取り、W&B ダッシュボード に ログ 記録されるテーブルの行に入力する簡単なユーティリティ関数を作成します。
wandb.Table オブジェクトと、データ可視化を取り込むための列(columns)を定義します。
train_dataset と val_dataset をそれぞれループして、データサンプルの可視化を生成し、 ダッシュボード に ログ 記録するテーブルの行を埋めます。
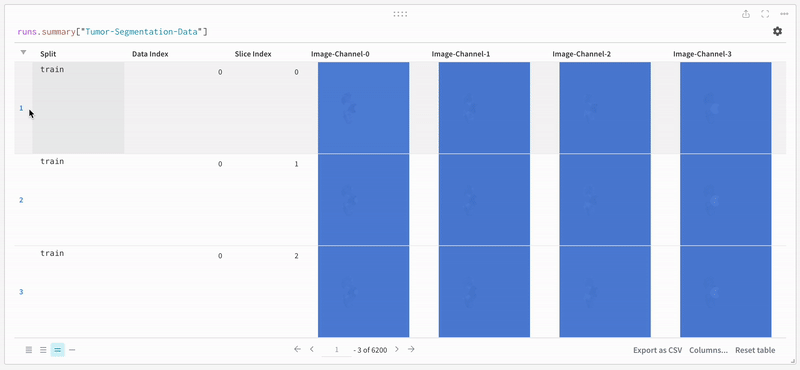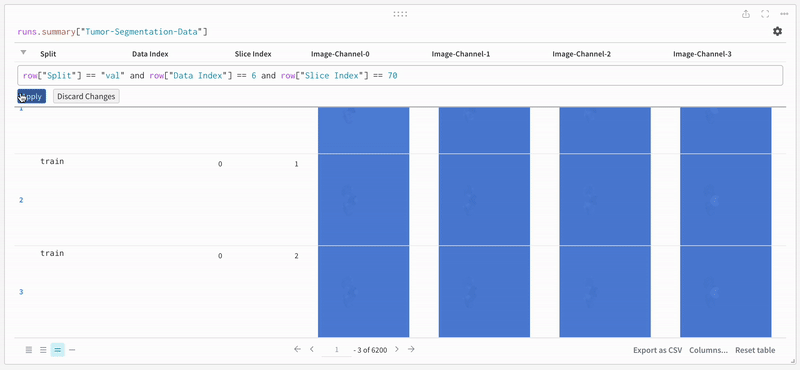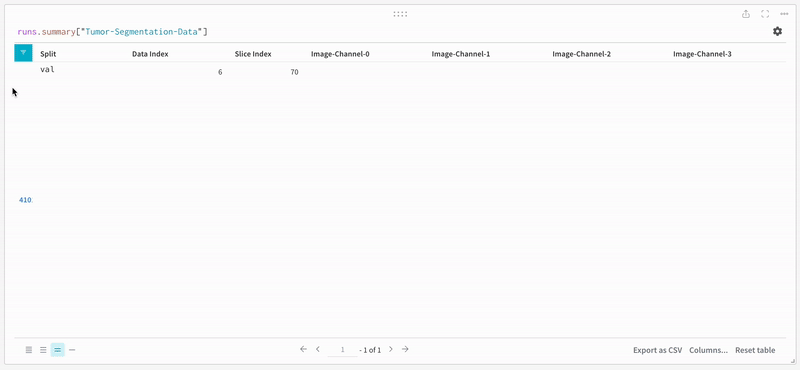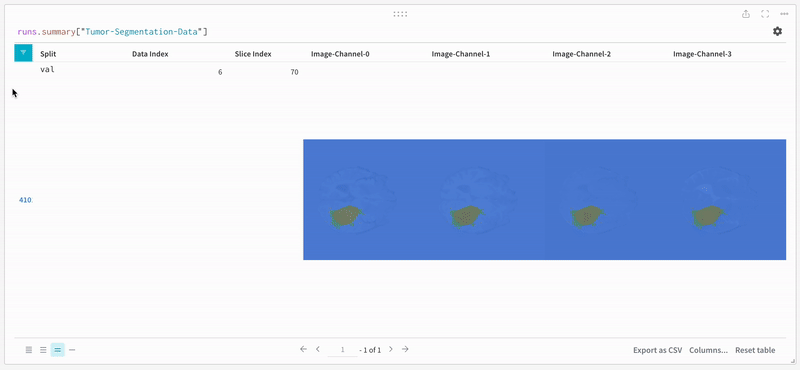
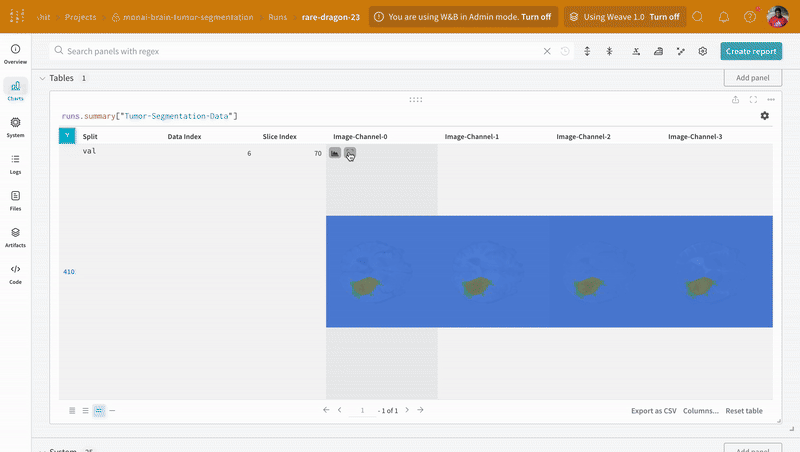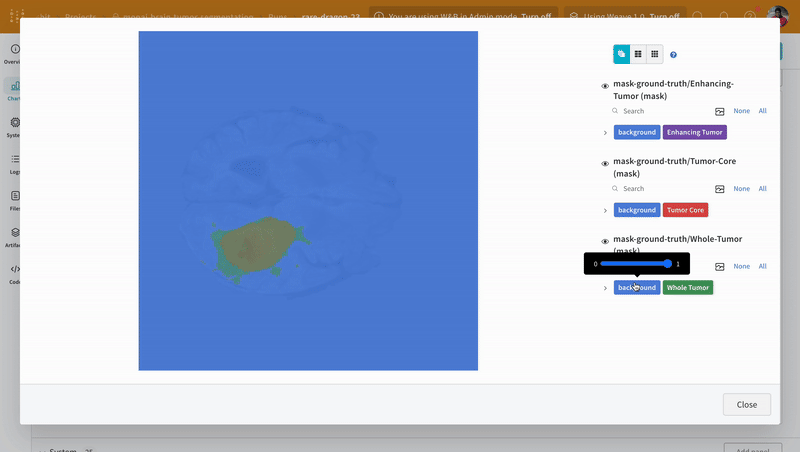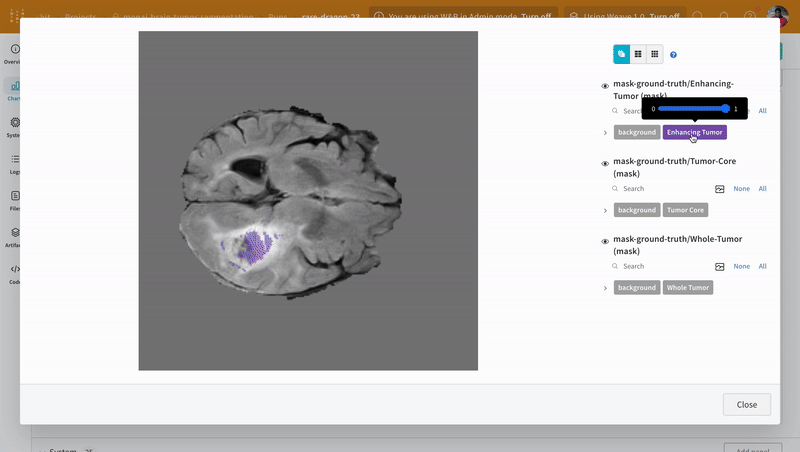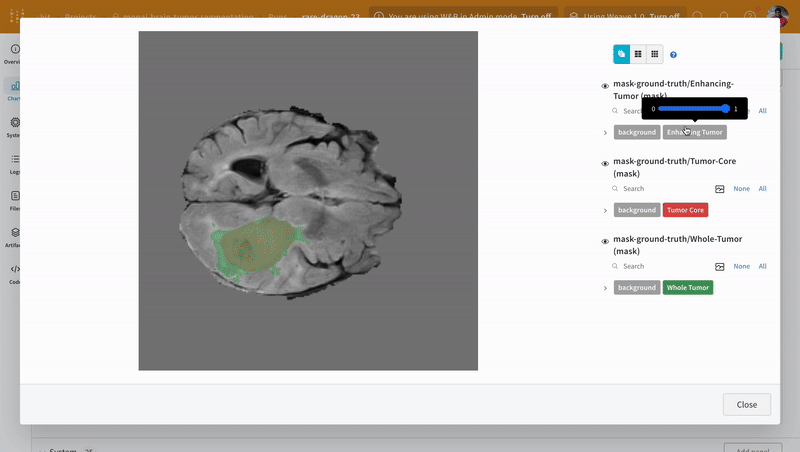
注: データセット のラベルは、クラス間で重複しないマスクで構成されています。オーバーレイでは、ラベルが個別のマスクとして ログ 記録されます。
データの読み込み
データセット からデータを読み込むための PyTorch DataLoader を作成します。 DataLoader を作成する前に、train_dataset の transform を train_transform に設定して、トレーニング用にデータを前処理および変換します。
モデル、損失関数、オプティマイザーの作成
このチュートリアルでは、論文 3D MRI brain tumor segmentation using auto-encoder regularization に基づいたSegResNet モデル を作成します。 SegResNet モデル は monai.networks API の一部として PyTorch モジュールとして実装されており、オプティマイザー や学習率スケジューラも含まれています。
monai.losses API を使用してマルチラベル DiceLoss として定義し、対応する Dice メトリクス を monai.metrics API を使用して定義します。
トレーニングと検証
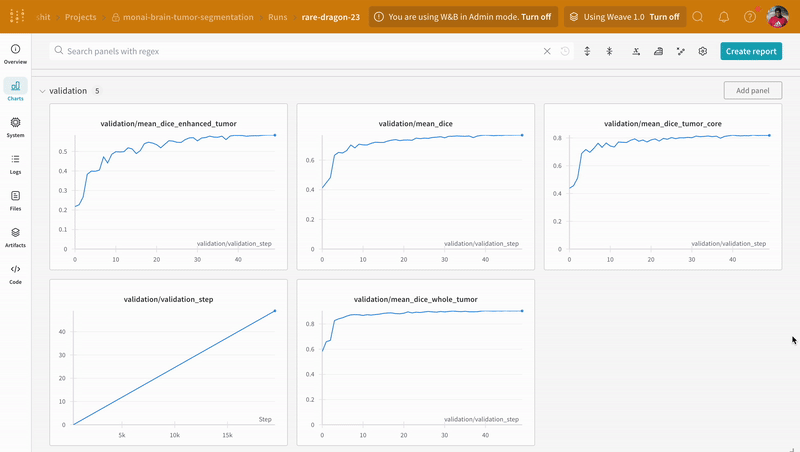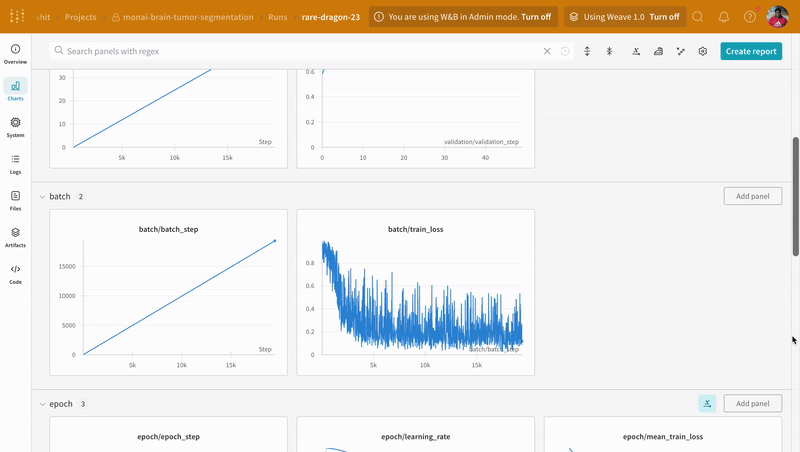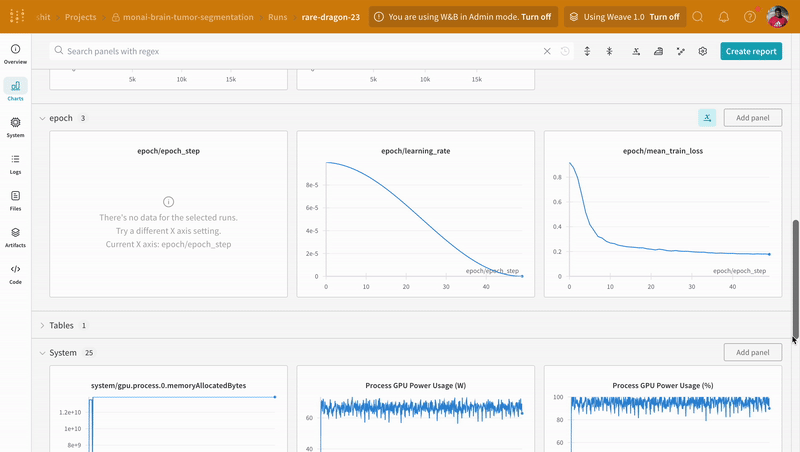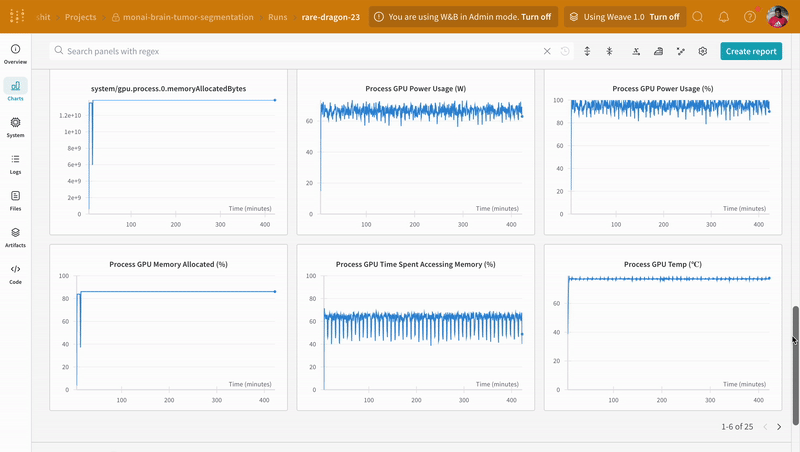
トレーニングの前に、トレーニングと検証の 実験 を追跡するためにrun.log() で後ほど ログ 記録する メトリクス プロパティを定義します。
標準的な PyTorch トレーニングループの実行
wandb.log を組み込むことで、トレーニングおよび検証 プロセス に関連するすべての メトリクス を追跡できるだけでなく、すべてのシステム メトリクス (この場合は CPU と GPU )も W&B ダッシュボード 上で追跡できるようになります。

推論
アーティファクトインターフェースを使用して、どの バージョン の アーティファクト が最適な モデル チェックポイントであるか(この場合は、エポックごとの平均トレーニング損失が最小のものなど)を選択できます。また、 アーティファクト の完全な リネージ (系統)を探索し、必要な バージョン を使用することもできます。
予測 の可視化と 正解 ラベルとの比較
学習済み モデル の 予測 を可視化し、インタラクティブなセグメンテーションマスクオーバーレイを使用して対応する 正解 セグメンテーションマスクと比較するための別のユーティリティ関数を作成します。